Mise en place§
Créez un environnement virtuel.
Installez-y
flaskavec la commande :pip install flask
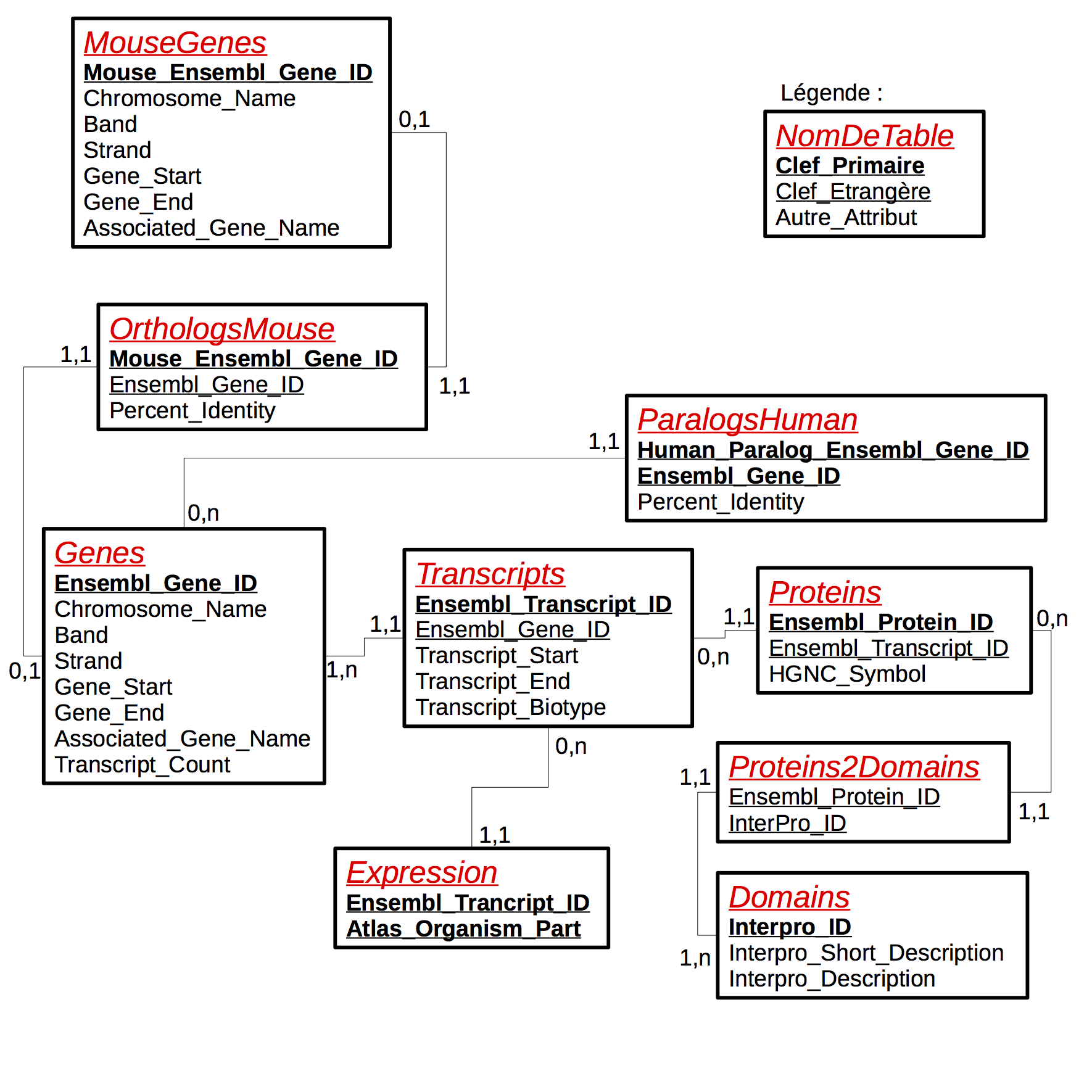
Téléchargez la base de données ensembl_hs63_simple.sqlite, dont le schéma relationnel vous est donné ci-dessous :
Cette base est un sous-ensemble de la base de données Ensembl. Il s'agit d'une base de données d’annotations de génomes contenant des informations sur les gènes, leurs transcrits, ainsi que de nombreux autres renseignements comme la fonction des gènes ou encore les comparaisons entre plusieurs génomes.
Votre travail§
Créez une application Flask répondant aux routes suivantes :
/- Une page de garde affichant la liste (triée par ordre alphabétique)
des
atlas_organism_partde la tableExpression. Chaque nom est un lien vers la liste de gènes correspondante (cf./parts/<part>/genesci-dessous). /parts/<part>/genesUne page affichant les identifiants (
ensembl_gene_id) et les noms (associated_gene_name) des gènes associées à cette partie d'organisme. Notez que l'association se fait via la tableTranscript; voici une requête SQL permettant de récupérer cette information⋅:SELECT DISTINCT g.ensembl_gene_id, associated_gene_name FROM Genes as g NATURAL JOIN Transcripts as t NATURAL JOIN Expression as e WHERE atlas_organism_part = ? ORDER BY g.ensembl_gene_id
L'identifiant et le nom de chaque gène doivent être des liens vers la fiche individuelle de ce gène (cf.
/genes/<id>ci-dessous)./genes/<id>La fiche individuelle du gène ayant l'identifiant
id.Elle doit contenir
- une section présentant toutes les informations données par ligne correspondante de la table
Genes, - une section présentant la liste des transcrits de ce gène (avec leur identifiant, et leurs position de début et de fin),
- une section avec la liste des parties d'organismes reliés (via les transcrits)
à ce gène (avec un lien vers la page
/parts/<part>/genescorrespondante.
- une section présentant toutes les informations données par ligne correspondante de la table
Utilisez les templates pour avoir une mise en page homogène sur toutes les pages du site, avec une feuille de style définie comme une ressource statique.
Pour aller plus loin§
Vous pouvez augmenter votre site avec les routes suivantes.
/genes/<id>/edit- un formulaire permettant de modifier un gène existant dans la base, accessible depuis la fiche individuelle du gène.
/transcripts/<id>une fichie individuelle de transcrit, affichant :
- toutes les informations données par ligne correspondante de la table
Transcripts, - un lien vers la fiche individuelle du gène correspondant à ce transcrit (
/genes/<id>), - une liste des parties d'organismes (avec des liens) associés à ce transcrit en particulier.
- toutes les informations données par ligne correspondante de la table